ABINIT-MP Openシリーズ (Ver.2 Rev.4)
※2020年6月版(Ver.1 Rev.22)に関するページはこちらです
※2020年6月版(Ver.1 Rev.22)に対応するBSV(BioStation Viewer)のWindows用インストーラはこちらからダウンロードできます
はじめに
フラグメント分子軌道(FMO)計算のプログラムABINIT-MP [1,2]のOpenシリーズは、2021年度にVer. 2系に移行しました。Ver. 2系では、これまで通りの機能の拡充に加え、常用されるメラープレセット2次摂動(MP2)レベルのジョブの「富岳」(理研)などの富士通A64FX系スーパーコンピュータでの数倍を睨んだ高速化、ならびに将来的には数万フラグメントの系を扱える大規模化対応が進められています。
A64FX向け高速化の第一段階として、SIMD(Single Instruction Multiple Data)化による2電子積分生成ルーチン群の改良が行われました。一方の大規模化対応では、フラグメント数の自乗的な配列を「FMO計算に本質的に必要なもの」のみに絞り込み、1万フラグメントの「壁」を超えました。この配列周りの改造に伴い、Ver. 1系までサポートされていた可視化インターフェースであるBSV(BioStation Viewer)での後解析のためのCPF(Check Point File)の書き出しを止めることになりました(BSV用にだけ保持する配列数が多数あるため)。標準的なスペックのWindows10 PCでは数千フラグメントの系のBSVでの可視化が困難であること、また分子動力学(MD)シミュレーションによって生成された多サンプル構造による統計的な相互作用エネルギー解析が増えてきていること、この2点もCPFのサポートを打ち切った判断の背景にはあります。
BSVを使った可視化的な解析については、今後も併存的に提供するVer. 1 Rev. 22を使っていただくことになりますが、Ver. 2系で入っていく新機能や種々の改良は今後とも反映されないことをお断りしておきます。
[1]“Electron-correlated fragment-molecular-orbital calculations for biomolecular and nano systems”, S. Tanaka, Y. Mochizuki, Y. Komeiji, Y. Okiyama, K. Fukuzawa, Phys. Chem. Chem. Phys. 16 (2014) 10310-10344.
[2] “The ABINIT-MP Program”, Y. Mochizuki, T. Nakano, K. Sakakura, Y. Okiyama, H. Watanabe, K. Kato, Y. Akinaga, S. Sato, J. Yamamoto, K. Yamashita, T. Murase, T. Ishikawa, Y. Komeiji, Y. Kato, N. Watanabe, T. Tsukamoto, H. Mori, K. Okuwaki, S. Tanaka, A. Kato, C. Watanabe, K. Fukuzawa (pp.53-67) in “Recent Advances of the Fragment Molecular Orbital Method – Enhanced Performance and Applicability”, ed. by Y. Mochizuki, S. Tanaka, K. Fukuzawa, (2021, Springer, Singapore).
Open Ver. 2 Rev. 4
Ver. 2系の最初のリリース版がRev. 4(2021年8月)です。機能的には、CPHF(Coupled-Perturbed Hartree-Fock)/LR(Linear Response)による動的分極率の算定[3]、フラグメント間相互作用エネルギー(IFIE: Inter-Fragment Interaction Energy)の機械学習用[4,5]の記述子データのダンプが可能となりました。分極率の評価は、機能性材料の解析や設計などで有用と思わます。図1に、Dodecaneのcc-pVDZ基底での動的分極率の計算例を示します。また、図2のchignolinのFMO-MP2/cc-pVDZの計算例のように記述子データはpython系ツールでの後処理に便利な形で出力されます。
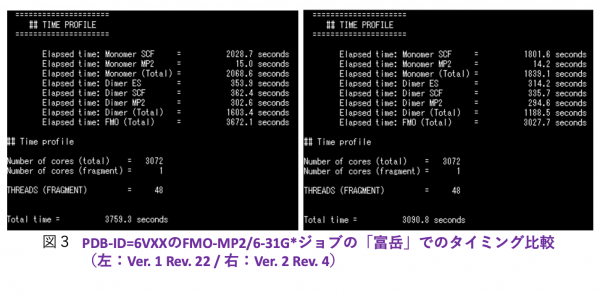
A64FX実行環境での高速化に関しては、2電子積分のSIMD化によってFMO-MP2計算では、Ver. 1 Rev. 2に比べて若干速くなっています(系とノード数にも依存)。図3に、新型コロナウイルスのスパイクタンパク質のクローズ構造PDB-ID=6VXX(3.3千残基)のFMO-MP2/6-31G*計算のVer. 1 Rev. 22とVer. 2 Rev. 4の「富岳」の8ラック、つまり3072ノード(48スレッド×3072プロセス)での時間比較を示します。ジョブ時間の短縮は62.7分→51.9分で、ステップ毎の比較では、モノマーSCC(Self-Consistent-Charge)条件を満たすまでフラグメントのHF(Hartree-Fock)計算が繰り返されるステップが速くなっていることが分かります。その他、細かな改良も施して加速を得ています。
大規模系への対応では、図4に示すインフルエンザウイルスのヘマグルチニンとFab抗体2つの複合体PDB-ID=1KENの水和モデル(MDで構造を生成)、総数で1.1万フラグメント(水とイオンが約9千個)の系のFMO-MP2/6-31G*ジョブが「富岳」の1ラックでは5.3時間で完走します。
[3] “Dynamic polarizability calculation with fragment molecular orbital scheme”, Y. Mochizuki, T. Ishikawa, K. Tanaka, H. Tokiwa, T. Nakano, S. Tanaka, Chem. Phys. Lett. 418 (2006) 418-422.
[4] “フラグメント分子軌道(FMO)計算の結果の自動解析の試み”, 望月祐志, 奥沢明, 計算工学学会誌, 22 (2017) 3539-3542.
[5] “FMOプログラムABINIT-MPの開発状況と機械学習との連携”, 望月祐志, 坂倉耕太, 秋永宜伸, 加藤幸一郎, 渡邊啓正, 沖山佳生, 中野達也, 古明地勇人,奥沢明, 福澤薫, 田中成典, J. Comp. Chem. Jpn., 16 (2017) 119-122.
Open Ver. 2 Rev. 4 (2021年8月版)の開発者
Ver. 2系の整備・開発は、下記のメンバで進められています(2021年8月時点)。
望月祐志*(立教大学 理学部), 中野達也(国立医薬品食品衛生研究所 医薬安全科学部), 坂倉耕太(計算科学振興財団), 渡邊啓正(HPCシステムズ), 佐藤伸哉(NECソリューションイノベータ), 奥脇弘次(立教大学 理学部)(*取り纏め責任者)
Open Ver. 2 Rev. 4のHPCI拠点への整備
Ver, 2 Rev. 4系は2021年9月から「富岳」、「不老 type I」(名古屋大学)、「Wisteria/Odyssey」(東京大学)のA64FXスーパーコンピュータに順次ライブラリプログラムとして順次登録されていきます。Ver. 1 Rev. 22も併存提供となります。また、実行バイナリには、Zn(II)含有タンパク質の6-31G*基底での計算のようにf型基底関数を要する系に対応する版が通常版と別に用意されます(バイナリのサイズも異なる)[6]。
「SQUIDベクトルノード群」(大阪大学)と「AOBA-A」(東北大学)のNEC SX-Aurora TSUBASAのベクトル型スーパーコンピュータでは、ベクトル化チューニングが施されたVer. 1 Rev. 22が整備されています。このチューニングによって、新型コロナウイルスのメインプロテアーゼとN3リガンドの複合体PDB-ID=6LU7のFMO-MP2/6-31G*計算では4.6倍の高速化が達成されています。
他に、「ITO Subsystem A」(九州大学)、「TSUBAME」(東京工業大学)、「Grand Chariot」(北海道大学)、「Wisteria/Aquarius」のIntel Xeon系スーパーコンピュータにもVer. 2 Rev. 4とVer. 1 Rev. 22をライブラリプログラムとして2021年内に整備見込みです。
Intel Xeon Phi(Kinghts Landing)を積む「Oakforest-PAICS」(JCAHPC)は2022年3月での退役が近いですが、ハイパースレッディングによって少数ノードで効率よくFMO-MP2計算が実行[7]できますので、Ver. 2 Rev. 4とVer. 1 Rev. 22のライブラリ整備を行います(2021年10月を予定)。
[6] “FMOプログラムABINIT-MPの整備状況2020”, 望月祐志, 坂倉耕太, 渡邊啓正, 奥脇弘次, 加藤幸一郎, 渡辺尚貴, 沖山佳生, 福澤薫, 中野達也, J. Comp. Chem. Jpn., 19 (2020) 142-145.
[7] “FMOプログラムABINIT-MPのOakForest-PACS上での多層並列化と性能評価”, 渡邊啓正, 佐藤伸哉, 坂倉耕太, 齊藤天菜, 望月祐志, J. Comp. Chem. Jpn., 17 (2018) 147-149.
Open Ver.2系の今後のリリース
Open Ver. 2系では、Ver. 1系までの生物物理・創薬系での応用重視から一般の化学分野での利用を強く意識した機能強化が図られる予定です。
Ver. 2の次のリリース版となるRev. 8では、IFIEの成分分析(PIEDA)[8,9]にLRD(Local Response Dispersion)機能[10]が入り、単一ジョブでの処理が可能となります。LRDもOpenMP/MPIの混成並列に対応します。通常のPIEDAでは、相関エネルギー補正を“DI”(分散:Dispersionの意)として括っているのですが、実際には分散力系の安定化エネルギーだけではなく、過剰なイオン性の低減などの電子相関の導入によるエネルギー低下も含まれており、個々のフラグメントの組み合わせによって両者の割合は変わります[11]。LRDではHF電子密度の多重極展開によって分散力系の安定化だけを算定しますので、“DI”の内訳を分けて解析できます。2021年8月時点で機能テスト中となっています。
長年の懸案となっている、多層FMO(MFMO)[12]によるCIS(Configuration Interaction Singles)系の励起エネルギーの算定機能[13-16]もRev. 8で「復活」します(2021年度下期に作業予定)。また、スケーリング補正付の2次のグリーン関数によるイオン化ポテンシャルの計算[17]もできるようになります。
Rev. 8での高速化と大規模系への対応は、FMO-MP2/6-31G*ジョブを基準に「可能な限りの向上」を図ることになりそうです。
Rev. 8の次はRev. 12を想定しており、これまでのMCP(Model Core Potential)[18]だけでなく、石村和也氏のSMASH[19]からのECP(Effective Core Potential)モジュールの移植によりLANL2DZ[20-22]の使用を可能とする予定です。ECPの方がカバーしている重元素が多く、GAUSSIANの結果との比較も直截です。
[8] “Pair Interaction Energy Decomposition Analysis”, D. G. Fedorov, K. Kitaura, J. Comp. Chem. 28 (2007) 222-237.
[9] “相互作用エネルギー成分分割解析機能PIEDAの実装とタンパク質-リガンド間の相互作用解析”, 塚本貴志, 加藤幸一郎, 加藤昭史, 中野達也, 望月祐志, 福澤薫, J. Comp. Chem. Jpn., 14 (2015) 1-9.
[10] “Density functional method including weak interactions: Dispersion coefficients based on the local response approximation”, T. Sato, H. Nakai, J. Chem. Phys. 131 (2009) 224104-1-12.
[11] “Fragment Molecular Orbital Based Interaction Analyses on Complexes Between SARS-CoV-2 RBD Variants and ACE2”, K. Akisawa, R. Hatada, K. Okuwaki, S. Kitahara, Y. Tachino, Y. Mochizuki, Y. Komeiji, S. Tanaka, Jpn. J. Appl. Phys. 60 (2021) 090901-1-5.
[12] “Multilayer Formulation of the Fragment Molecular Orbital Method (FMO)”, D. G. Fedorov, T. Ishida, K. Kitaura, J. Phys. Chem. A, 109 (2005) 2638-2646.
[13] “Configuration interaction singles method with multilayer fragment molecular orbital scheme”, Y. Mochizuki, S. Koikegami, S. Amari, K. Segawa, K. Kitaura, T. Nakano, Chem. Phys. Lett. 406 (2005) 283-288.
[14] “Parallelized integral-direct CIS(D) calculations with multilayer fragment molecular orbital scheme”, Y. Mochizuki, K. Tanaka, K. Yamashita, T. Ishikawa, T. Nakano, S. Amari, K. Segawa, T. Murase, H. Tokiwa, M. Sakurai, Theor. Chem. Acc. 117 (2007) 541-553.
[15] “Modification for spin-adapted version of configuration interaction singles with perturbative doubles”, Y. Mochizuki, K. Tanaka, Chem. Phys. Lett., 443 (2007) 389-397.
[16] “A practical use of self-energy shift for the description of orbital relaxation”, Y. Mochizuki, Chem. Phys. Lett., 453 (2008) 109-116.
[17] J. Linderberg, Y. Öhrn, “Propagators in Quantum Chemistry, Second Edition”, (2004, Wiley, Hoboken NJ).
[18] “Fragment molecular orbital calculations on large scale systems containing heavy metal atom”, T. Ishikawa, Y. Mochizuki, T. Nakano, S. Amari, H. Mori, H. Honda, T. Fujita, H. Tokiwa, S. Tanaka, Y. Komeiji, K. Fukuzawa, K. Tanaka, E. Miyoshi, Chem. Phys. Lett., 427 (2006) 159-165.
[19] https://sourceforge.net/projects/smash-qc/
[20] “Ab initio effective core potentials for molecular calculations. Potentials for the transition metal atoms Sc to Hg”, P. J. Hay, W. R. Watt, J. Chem. Phys. 82 (1985) 270-283.
[21] “Ab initio effective core potentials for molecular calculations. Potentials for main group elements Na to Bi”, W. R. Watt, P. J. Hay, J. Chem. Phys. 82 (1985) 284-298.
[22] “Ab initio effective core potentials for molecular calculations. Potentials for K to Au including the outermost core orbitals”, P. J. Hay, W. R. Watt, J. Chem. Phys. 82 (1985) 299-310.
謝辞
ABINIT-MP Open Ver. 2系のA64FX向けの高速化と大規模系への対応は、学際大規模情報基盤共同利用・共同研究拠点(JHPCN)の2021年度課題jh210036-NAH「FMOプログラムABINIT-MPの高速化と超大規模系 への対応」(代表者:望月祐志)の中で展開されており、A64FXスーパーコンピュータ「不老」を備える名古屋大学情報基盤センターの片桐孝洋・大島聡史先生の研究室とのコラボレーションで進行しています。また、富士通(株)様のサイエンティフィック・システム研究会(SS研)の「A64FXシステムアプリ性能検討WG」の活動とも連動しており、種々のサポートをいただいています。
「富岳」でのテスト計算は、2021年度のHPCI課題hp210026「新規感染症のための計算科学的解析環境の整備」(代表者:望月祐志)の中で行っています。
NEC SX-Aurora TSUBASA向けのベクトル化チューニング、さらに解析機能向上については日本電気(株)様からご支援をいただいています。また、数社様からの立教大学への寄付金を得て、2020~2021年度のABINIT-MPプログラムの研究開発と維持ができています。
HPCI拠点へのABINIT-MPのライブラリ整備やハンズオンセミナーでお世話になっている(一財)高度情報科学技術研究機構(RIST)に謝意を表します。
最後に、「計算工学ナビ」サイトでのABINIT-MPの情報公開で長年ご支援いただいている東京大学生産技術研究所の加藤千幸先生、同革新的シミュレーション研究センターにも感謝したいと思います。
コンタクト
ABINIT-MPのOpenシリーズの個別のご利用、あるいはプログラム開発にご関心のある方は、立教大学の望月祐志(fullmoon -at- rikkyo.ac.jp)にメールにてご連絡いただければ対応させていただきます(-at-を@に変換してください)。