ABINIT-MP Openシリーズ (Ver.1 Rev.22)
※2019年3月版(Ver.1 Rev.15)に関するページはこちらです
はじめに
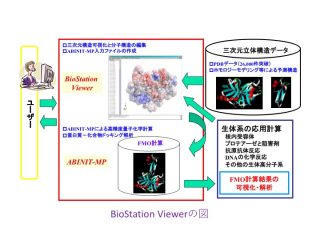
ABINIT-MPは、フラグメント分子軌道(FMO)計算を高速に行えるソフトウェアです[1]。専用GUIのBioStation Viewerとの連携により、入力データの作成~計算結果の解析が容易に行えます。4体フラグメント展開(FMO4)による2次摂動計算も可能です。また、部分構造最適化や分子動力学の機能もあります。FMOエネルギー計算では、小規模のサーバから超並列スパコンまで対応しています(Flat MPIとOpenMP/MPI混成)。
[1]“Electron-correlated fragment-molecular-orbital calculations for biomolecular and nano systems”, S. Tanaka, Y. Mochizuki, Y. Komeiji, Y. Okiyama, K. Fukuzawa, Phys. Chem. Chem. Phys. 16 (2014) 10310-10344.
特徴
ABINIT-MPは使い易いFMOプログラムで、4体フラグメント展開までが可能です。研究室単位のLinux/Intel系サーバに標準搭載されているMPI環境で動作しますし、特別な設定も必要ありません。また、煩雑で注意深さを要するフラグメント分割を伴う入力データの作成は随伴GUIのBioStation Viewer(Windowsで動作)を使うなどすれば容易に作成出来ます。また、フラグメント間相互作用エネルギー(IFIE)などの計算結果は膨大となりプリントからの理解はしばしば困難ですが、Viewerを使うと可視的・直観的に対象系の相互作用の様態を把握出来ます。
開発の経緯
ABINIT-MPプログラムは、東京大学生産技術研究所を拠点とする文科省系の「戦略的基盤ソフトウェアの開発」、「革新的シミュレーションソフトウェアの開発」、「HPCI戦略分野4 次世代ものづくり」の一連のプロジェクト(代表:東京大学 加藤千幸教授)、さらにJST-CRESTの「シミュレーション技術の革新と実用化基盤の構築」(代表:神戸大学 田中成典教授)と立教大学SFR(担当:望月祐志)などの支援を得て、約20年に渡って開発が進められてきました。Intel Xeon (IA64)系バイナリは、Ver. 7が東京大学生産技術研究所の革新的シミュレーション研究センター[2]で利用可能となっています(2020年3月時点)。
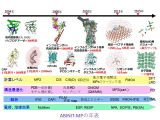
2015年度からは、東京大学工学研究科を代表拠点とする文科省プロジェクト「フラッグシップ2020 重点課題6」(代表:東京大学 吉村忍教授)[3]の中で、Openシリーズとして機能強化・高速化とリリースが行われてきました(取り纏め責任者:立教大学 望月祐志)。第一版は、バイナリで公開してきているVer. 7を元にメモリー要求の軽減などの改良を施したもので、2016年12月にまとまったVer. 1 Rev. 5 [4]です。Rev. 5は、「京」など幾つかのHPCI拠点にライブラリとして提供されました。
Ver. 1 Rev. 5をベースとし、成分毎の相互作用エネルギー解析(PIEDA)[5]、局在化軌道による分散力の局所解析(FILM)[6]、電荷移動の軌道対解析(CAFI)[7]のなどの解析系の機能強化を図った版が2018年2月リリースのVer. 1 Rev. 10 [8]です。東京大学・筑波大学のJCAHPC管理のメニーコアCPU機であるOakForest-PACS向けには、利用頻度の高いMP2エネルギー計算でOpenMP/MPI/MPIの3階層の並列化実行も可能となりました。なお、2018年の秋から専用GUI であるBioStation Viewerの更新・整備の管轄者は星薬科大学の福澤薫准教授に移りました。
2019年3月にリリースしたVer. 1 Rev. 15 [8]では、ペプチド結合部分のカルボニル酸素(>C=O)による相互作用で、しばしば問題になってきた「相互作用フラグメントの帰属ズレ問題」[9]に対する解決策の一つとして、部分3体展開下でsp2混成炭素(BDA)でのフラグメント切断が可能となりました。エネルギー微分にも対応しています[13]。もう一つの新規機能は、局所応答分散(LRD)[10]によってMP2に代わって分散力による安定化エネルギーを算定出来るようになったことで(論文投稿を準備中)、計算コストとメモリー要求がMP2に比して低いので大きな系を扱うのに有利です。また、結合クラスター展開(CCSD(T)も可)と多体摂動(MBPT)のモジュール[11]が旧JST-CREST版から移植されました。その他、ポアソンボルツマン(PB)型の水和[12]でキャビティを定義する有効原子半径の汎用性を上げています。
「フラッグシップ2020 重点課題6」の枠組みの中での最終版となったのが、Ver. 1 Rev. 20(2020年3月)です。この版では、PB水和での表面領域モデル(PBSA)[14]とPB条件下でのPIEDAの計算がサポートされました。さらに、FMO多層近似[15]によってタンパク質・リガンド複合体の全体ではなく、重要領域のファーマコフォアのみに3次までの摂動(MP3)計算[16]を行い、経験的スケーリング(MP2.5)によってCCSD(T)に近い相互作用エネルギー[17]を得ることも可能となりました。もう1つ追加の機能向上は、基底関数にf型が使えるようになったことで、Zn(II)を含むタンパク質の計算も6-31G*基底で行えます(エネルギー微分も可能)。
Rev. 20に対し、PIEDA処理を大幅に高速化した版が2020年6月リリースのRev. 22です。この緊急改良は、2020年4月からの「富岳」(旧名はポスト「京」)の早期利用による新型コロナウイルスの対策プロジェクト[18]の中、3.3千残基のスパイクタンパク質の計算を行う際に必要となりましたが、結果としてオーバーヘッドは解消されました。最近の応用計算ではPIEDAが常用されていますので、この更新は大きな価値を持つと考え、Rev. 22を最新リリースの対象としました。
HPCI拠点でのライブラリプログラムとしての登録は、高度情報科学技術研究機構(RIST)のご支援を2018年度の下期から受けており、これまでのVer. 1のRev. 10やRev. 15を整備していただいています。Rev. 22以降もこうしたご支援をいただける見込みです。
[2] http://www.ciss.iis.u-tokyo.ac.jp/
[3] http://postk6.t.u-tokyo.ac.jp/
[4] “FMOプログラムABINIT-MPの開発状況と機械学習との連携”, 望月祐志, 坂倉耕太, 秋永宜伸, 加藤幸一郎, 渡邊啓正, 沖山佳生, 中野達也, 古明地勇人,奥沢明, 福澤薫, 田中成典, J. Comp. Chem. Jpn., 16 (2017) 119.
[5] “Pair Interaction Energy Decomposition Analysis”, D. G. Fedorov, K. Kitaura, J. Comp. Chem. 28 (2007) 222.
[6] “Fragment interaction analysis based on local MP2”, T. Ishikawa, Y. Mochizuki, S. Amari, T. Nakano, H. Tokiwa, S. Tanaka, K. Tanaka, Theor. Chem. Acc. 118 (2007) 937.
[7] “A configuration analysis for fragment interaction”, Y. Mochizuki, K. Fukuzawa, A. Kato, S. Tanaka, K. Kitaura, T. Nakano, Chem. Phys. Lett. 410 (2005) 247.
[8] “ABINIT-MP Openシリーズの最新の開発状況について”, 望月祐志, 秋永宜伸, 坂倉耕太, 渡邊啓正, 加藤幸一郎, 渡辺尚貴, 奥脇弘次, 中野達也, 福澤薫, J. Comp. Chem. Jpn., 18 (2019) 129.
[9] “Antigen-antibody interactions of influenza virus hemagglutinin revealed by the fragment molecular orbital calculation”, A. Yoshioka, K. Takematsu, I. Kurisaki, K. Fukuzawa, Y. Mochizuki, T. Nakano, E. Nobusawa, K. Nakajima, S. Tanaka, Theor. Chem. Acc. 130 (2011) 1197.
[10] “Density functional method including weak interactions: Dispersion coefficients based on the local response approximation”, T. Sato, H. Nakai, J. Chem. Phys. 131 (2009) 224104-1.
[11] “Higher-order correlated calculations based on fragment molecular orbital scheme”, Y. Mochizuki, K. Yamashita, T. Nakano, Y. Okiyama, K. Fukuzawa, N. Taguchi, S. Tanaka, Theor. Chem. Acc. 130 (2011) 515.
[12] “Fragment Molecular Orbital Calculations with Implicit Solvent Based on the Poisson-Boltzmann Equation: Implementation and DNA Study”, Y. Okiyama, T. Nakano, C. Watanabe, K. Fukuzawa, Y. Mochiuzki, S. Tanaka, J. Phys. Chem. B 122 (2018) 4457.
[13] “Fragmentation at sp2 carbon in fragment molecular orbital (FMO) method”, Y. Akinaga, K. Kato, T. Nakano, K. Fukuzawa, Y. Mochizuki, J. Comp. Chem., 41 (2020) 1416.
[14] “Fragment Molecular Orbital Calculations with Implicit Solvent Based on the Poisson-Boltzmann Equation: II. Protein and Its Ligand-Binding System Studies”, Y. Okiyama, C. Watanabe, K. Fukuzawa, Y. Mochiuzki, T. Nakano, S. Tanaka, J. Phys. Chem. B, 123 (2019) 957.
[15] “Multilayer Formulation of the Fragment Molecular Orbital Method (FMO)”, D. G. Fedorov, T. Ishida, K. Kitaura, J. Phys. Chem. A, 109 (2005) 2638.
[16] “Large-scale FMO-MP3 calculations on the surface proteins of influenza virus, hemagglutinin (HA) and neuraminidase (NA)”, Y. Mochizuki, K. Yamashita, K. Fukuzawa, K. Takematsu, H. Watanabe, N. Taguchi, Y. Okiyama, M. Tsuboi, T. Nakano, S. Tanaka, Chem. Phys. Lett., 493 (2010) 346.
[17] “Fragment molecular orbital (FMO) calculations on DNA by a scaled third-order Moeller-Plesset perturbation (MP2.5) scheme”, H. Yamada, Y. Mochizuki, K. Fukuzawa, Y. Okiyama, and Y. Komeiji, Comp. Theor. Chem. 1101 (2017) 46.
[18] https://www.r-ccs.riken.jp/library/topics/fugaku-coronavirus.html
Open Ver. 1 Rev. 22 (2020年6月版)の主な機能
Open Ver. 1 Rev. 22では、下記の機能が利用可能となっています。
- エネルギー
→ FMO4: HF, MP2 (CD)
→ FMO2:HF, LRD, MP2, MP3, CC/MBPT
- エネルギー微分
→ FMO4: HF, MP2
→ FMO2: MP2構造最適化(凍結領域可), MD
- その他機能
→ SCIFIE, CAFI, 重要データ書出し(CPF, IDF)
→ MCP, PB水和, BSSE-CP
→ PIEDA, FILM
→ sp2炭素での切断 (部分FMO3)
→ GUI(BioStation Viewer)
- 並列化環境(PC~スパコン)
→ MPI, OpenMP/MPI混成
→ MP2エネルギーはOpenMP/MPI/MPIの3階層混成
→ 最深部はBLAS処理 (DGEMM)
Open Ver. 1 Rev. 22 (2020年6月版)の開発者
望月祐志*(立教大学 理学部), 中野達也(国立医薬品食品衛生研究所 医薬安全科学部), 坂倉耕太(FOCUS), 沖山佳生(国立医薬品食品衛生研究所 医薬安全科学部), 渡邊啓正(HPCシステムズ), 加藤幸一郎(九州大学), 渡辺尚貴(みずほ情報総研), 秋永宜伸(ヴァイナス), 佐藤伸哉(NECソリューションイノベータ), 山本純一(NECソリューションイノベータ), 山下勝美(元 NECソフト), 村瀬匡(元 NECソフト), 石川岳志(鹿児島大学 学術研究院 理工学域工学系), 古明地勇人(産業技術総合研究所 バイオシステム部門), 加藤雄司(元 立教大学 理学部), 塚本貴志(みずほ情報総研), 森寛敏(中央大学 理工学部), 奥脇弘次(立教大学 理学部), 田中成典(神戸大学大学院 システム情報学研究科), 加藤昭史(スコーピオンテック), 渡邉千鶴(理研 横浜), 福澤薫**(星薬科大学 薬学部) (*ABINIT-MP取り纏め責任者 / **BioStation Viewer責任者)
応用分野
ABINIT-MPのFMO計算は、開発当初から生体分子関係、特にタンパク質とリガンド(薬品分子)の複合系に対して主に用いられてきました。これは、計算で得られるフラグメント間相互作用エネルギーがアミノ酸残基間、あるいはリガンド-アミノ酸残基間の相互作用の状態を理解するのに好適なためです[1]。しかし、FMO計算は生体系に限られるだけでなく、水和凝集系や一般の高分子、あるいは固体[19-21]なども扱える潜在力を持っています。「フラッグシップ2020 重点課題6」の研究開発活動の中では、有効相互作用パラメータをFMOで求めて高分子の粗視化シミュレーションを行うマルチスケール計算手法と応用が進められ[22-24]、さらに脂質膜[25]やタンパク質[26]にも適用されました。
ABINIT-MPの利活用が、今後こうした一般の化学工学や材料科学、あるいは応用物理関係の分野へも広がっていくことを期待していますし、そのための整備とエビデンスの集積を推進していきます。また、多構造サンプルの計算結果を統計的に解析する試み(機械学習も適宜利用)も始まっています[27,28]。
[19] “Modeling of peptide – silica interaction based on four-body corrected fragment molecular orbital (FMO4) calculations”, Y. Okiyama, T. Tsukamoto, C. Watanabe, K. Fukuzawa, S. Tanaka, Y. Mochizuki, Chem. Phys. Lett., 566 (2013) 25.
[20] “Modeling of hydroxyapatite – peptide interaction based on fragment molecular orbital method”, K. Kato, K. Fukuzawa, Y. Mochizuki, Chem. Phys. Lett., 629 (2015) 58.
[21] “Interaction between calcite and adsorptive peptide analyzed by fragment molecular orbital method”, K. Kato, K. Fukuzawa, Y. Mochizuki, Jpn. J. Appl. Phys., 58 (2019) 120906.
[22] “Fragment Molecular Orbital-based Parameterization Procedure for Mesoscopic Structure Prediction of Polymeric Materials”, K. Okuwaki, Y. Mochizuki, H. Doi, T. Ozawa, J. Phys. Chem. B 122 (2018) 338.
[23] “フラグメント分子軌道(FMO)法を用いた散逸粒子動力学シミュレーションのための有効相互作用パラメータ算出の自動化フレームワーク”, 奥脇弘次, 土居英男, 望月祐志, J. Comp. Chem. Jpn. 17 (2018) 102.
[24] “Theoretical Analyses on Water Cluster Structures in Polymer Electrolyte Membrane by Using Dissipative Particle Dynamics Simulations with Fragment Molecular Orbital Based Effective Parameters”, K. Okuwaki, Y. Mochizuki, H. Doi, S. Kawada, T. Ozawa, K. Yasuoka, RSC Adv. 8 (2018) 34582.
[25] “Dissipative particle dynamics (DPD) simulations with fragment molecular orbital (FMO) based effective parameters for 1-Palmitoyl-2-oleoyl phosphatidyl choline (POPC) membrane”, H. Doi, K. Okuwaki, Y. Mochizuki, T. Ozawa, K. Yasuoka, Chem. Phys. Lett. 684 (2017) 427.
[26] “Folding simulation of small proteins by dissipative particle dynamics (DPD) with non-empirical interaction parameters based on fragment molecular orbital calculations”, K. Okuwaki, H. Doi, K. Fukuzawa, Y. Mochizuki, Appl. Phys. Express 13 (2020) 017002.
[27] “Destabilization of DNA through interstrand crosslinking by UO22+”, A. Rossberg, T. Abe, K. Okuwaki, A. Barkleit, K. Fukuzawa, T. Nakano, Y. Mochizuki, S. Tsushima, Chem. Comm. 55 (2019) 2015.
[28] “Cm3+/Eu3+ Induced Structural, Mechanistic and Functional Implications for Calmodulin”, B. Drobot, M. Schmidt, Y. Mochizuki, T. Abe, K. Okuwaki, F. Brulfert, S. Falke, S. A. Samsonov, Y. Komeiji, C. Betzel, T. Stumpf, J. Raff, S. Tsushima, Phys. Chem. Chem. Phys. 21 (2019) 21213.
Openシリーズの今後のリリース
ABINIT-MPには、主に開発の経緯的な事由から「ローカル版」が存在しています[1]。Openシリーズでは、Ver. 1 Rev. 20まで統合を図ってきましたが、励起エネルギー[29,30]や動的分極率[31]の算定については整備計画の変更もあり、残念ながら公開に至りませんでした。「フラッグシップ2020 重点課題6」は2019年度(2020年3月)で終了していますが、今後もリリースを続けていきたいと考えています。Ver. 1系の最終版の位置づけとなるRev. 25は2021年春の公開を目指しており、Rev. 22に上記の励起状態関係の機能、機械学習用データのダンプの機能、福澤准教授が参画されているAMED-BINDSプロジェクト[32]で整備中の凍結領域構造最適化[33]などが入る予定です。また、MP2エネルギー微分のベクトル化も対応予定です。
Ver. 2はVer. 1 Rev. 25をベースとしますが、P系では鹿児島大学の石川岳志教授のPAICS [34]から2電子積分の恒等分解(RI)に基づくRI-MP2/MP3モジュール(C言語で記述)が移植されて使えるようになります(ローカル版での作業は既に完了)。一方、S系では石村和也氏のSMASH [35]から提供いただいている積分モジュールやDFT機能を前面に出すことになります(LRDでの数値積分でDFTのルーチンは既に一部を利用)。また、超大規模系となる1万フラグメントまでの対応も検討しています。
OpenシリーズのHPCI拠点以外でのバイナリのオンデマンド的な提供は今後も継続し、以下のようなプラットフォームを対象(予定含む)とします(2020年6月時点)。
- PC: Windows 10 (64 bit)
- 小規模サーバ: Intel Xeon (IA64) & Xeon Phi (Knights Landing)
- スーパーコンピュータ: Intel Xeon系, 富士通-ARM系, NEC-SX系 (Vector)
[29] “Configuration interaction singles method with multilayer fragment molecular orbital scheme”, Y. Mochizuki, S. Koikegami, S. Amari, K. Segawa, K. Kitaura, and T. Nakano, Chem. Phys. Lett. 406 (2005) 283.
[30] “Parallelized integral-direct CIS(D) calculations with multilayer fragment molecular orbital scheme”, Y. Mochizuki, K. Tanaka, K. Yamashita, T. Ishikawa, T. Nakano, S. Amari, K. Segawa, T. Murase, H. Tokiwa, M. Sakurai, Theor. Chem. Acc. 117 (2007) 541.
[31] “Dynamic polarizability calculation with fragment molecular orbital scheme”, Y. Mochizuki, T. Ishikawa, K. Tanaka, H. Tokiwa, T. Nakano, S. Tanaka, Chem. Phys. Lett. 418 (2006) 418.
[33] “Geometry Optimization of the Active Site of a Large System with the Fragment Molecular Orbital Method”, D. G. Fedorov, Y. Alexeev, K. Kitaura, J. Phys. Chem. Lett. 2 (2011) 282.
[35] http://smash-qc.sourceforge.net/
FMO創薬コンソーシアム
2015年度から「FMO創薬コンソーシアム」(代表:星薬科大学 福澤薫 准教授)が産官学で組織され、「京」を計算資源としてABINIT-MPによるFMO計算基づくタンパク質・リガンドの相互作用解析が進められてきました[36](2020年度はOakForest-PACS上で活動)。重要な創薬ターゲットが設定されており、当該領域の共有基盤となる知見(特にIFIEのデータセット)が蓄積されています(IFIEはデータベース公開)[37]。ABINIT-MPはOpenシリーズとして改良が続けられていきますが、このコンソーシアムは実践的な利用者コミュニティとして重要な役割を果たしていくことになります。追記となりますが、BioStation Viewerはサイト[36]からダウンロード可能です。
[36] https://fmodd.jp/
[37] “Development of an automated fragment molecular orbital (FMO) calculation protocol toward construction of quantum mechanical calculation database for large biomolecules”, C. Watanabe, H. Watanabe, Y. Okiyama, D. Takaya, K. Fukuzawa, S. Tanaka, T. Honma, CBI-J. 19 (2019) 5.
開発系コンソーシアム
ABINIT-MPのOpenシリーズの開発や保守にソースレベルでコミットしていただくための産官学の枠組みです(コンタクト先:立教大学 望月祐志)。バグ情報と対策、新規開発の機能のシェアなど意図していますが、参画される企業様が商用に独自の高速化や改良を図ることは基本的に可とする方針です。現段階では、個別にご参画をお願い・確認させていただいており、分子科学研究所の藤田貴敏准教授からは励起状態計算機能[38,39]を提供いただくことになっています(リリースはVer. 2での予定)。その他にも、周期境界条件を課したFMOベースのMDの整備なども進んでいます。
[38] “Development of the Fragment Molecular Orbital Method for Calculating Non-local Excitations in Large Molecular Systems”, T. Fujita, Y. Mochizuki, J. Phys. Chem. A 122 (2018) 3886.
[39] “Development of the fragment-based COHSEX method for large and complex molecular systems”, T. Fujita, Y. Noguchi, Phys. Rev. B 98 (2018) 205140.
コンタクト
ABINIT-MPのOpenシリーズのご利用、あるいは開発系コンソーシアムにご関心のある方は、立教大学の望月祐志(fullmoon -at- rikkyo.ac.jp)にメールにてご連絡いただければと思います(-at-を@に変換してください)。ご所望の利用形態に応じて、個別に契約書面の取り交わしをさせていただき、ご提供したいと思います。よろしくお願いいたします。